Molekulare Zellbiologie
Aktuelle Forschungsthematik:
DNA-bindende Transkriptionsfaktoren sind entscheidende Schalter, um die verschiedensten Genexpressionsprogramme angepasst an die Erfordernisse einer Zelle in Zeit und Raum zu modulieren. Die größte Klasse humaner Transkriptionsfaktoren, mit etwa 750 Vertretern, wird durch die C2H2 Zinkfingerproteine (ZNF-Proteine) gebildet. Ungefähr die Hälfte von ihnen ist durch eine aminoterminale Protein-Protein Interaktionsdomäne gekennzeichnet, welche man Krüppel-assoziierte Box (Abkürzung KRAB) nennt.
Das kanonische Modell für die Transkriptionsregulation durch KRAB-ZNF-Proteine postuliert, dass diese Transkriptionsfaktoren über ihre Zinkfinger spezifisch an DNA binden und über ihre KRAB-Domäne Multiproteinkomplexe rekrutieren, welche letztendlich den erkannten Genlocus in einen für die Transkription nicht-permissiven Zustand überführen (Repression). Entscheidender Co-Faktor in diesem Modell ist das Multidömänenprotein TRIM28 (Synonym KAP1), welches als zentrale Plattform die Verbindung zu Proteinen mit Chromatin-modulierenden Aktivitäten herstellt. Wichtige Interaktionspartner sind vor allem Heterochromatinproteine (HP1), die Histon-H3-Lysin-9-Methyltransferase SETDB1 und der „nucleosome remodeling and deacetylase“ (NuRD) Komplex mit seiner Histondeacetylierungsaktivität.
Das KRAB-ZNF/TRIM28 System ist von Bedeutung in der Stammzellbiologie, bei Differenzierungsprozessen, bei der Antwort auf DNA-Schädigungen, der Unterdrückung von Retrotransposons und bei Tumorerkrankungen.
Die genauen Mechanismen wie ein KRAB-ZNF/TRIM28 Modul die biologischen Prozesse steuert, bedürfen weiterer Klärung. Offene Fragen betreffen die Steuerung des Moduls, insbesondere welche Komplexe wann und wie unter welchen biologischen Umständen (Zelltyp, Zellzyklus, endogene und exogene Stimuli) auf welche Art und Weise und auf welcher Ebene die Genregulation beeinflussen. Ebenso unbeantwortet sind die Funktionen der Mehrzahl der KRAB-ZNF Proteine, vor allem in Bezug auf die Identifizierung der von ihnen regulierten Zielgene. Evolutionär hat sich das TRIM28/KRAB-ZNF System vermutlich im Zuge der Entstehung der Landwirbeltiere (Tetrapoden) gebildet.
Die Aufklärung der Mechanismen des TRIM28/KRAB-ZNF Systems und die Charakterisierung von chemischen Verbindungen, welche dieses System beeinflussen, haben große Relevanz für das Verständnis und die Modulation programmierbarer Transrepressoren wie dCas9-KRAB (CRISPRi-Methodik) in der Gentechnik und Gentherapie. Dort wird die KRAB-Domäne des KRAB-ZNF Proteins Kox1/ZNF10 verwendet, um Gene zielgerichtet abzuschalten.
Methodische Ansätze:
- Anwendung von bioinformatischen Werkzeugen für phylogenetische Betrachtungen und Genexpressionsanalysen
- Verwendung von Knockout-Zelllinien zur Aufschlüsselung der Funktionalität von Komponenten der Genregulation
- Konstruktion von maßgeschneiderten eukaryotischen Expressionsvektoren für transiente und stabile Transfektion der verwendeten Zelllinien
- Aufklärung von Mechanismen der Transkriptionskontrolle mittels Reportergenassays
- Studium von Proteinkomplexen durch Co-Immunpräzipitation oder Affinitätschromatographie gefolgt von Massenspektrometrie (in Kooperation mit dem Proteomzentrum Rostock)
- Verfolgung von Genexpressionsprofilen mittels quantitativer Echtzeit-PCR
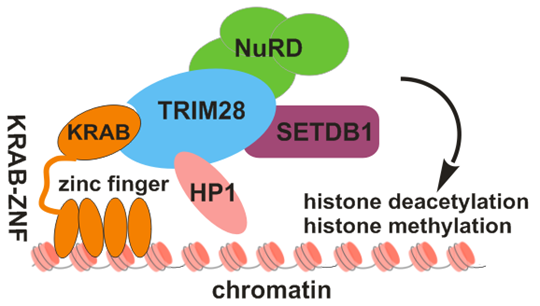
Abbildung: Modell für die transkriptionelle Regulation durch ein KRAB-Zinkfingerprotein
im Verbund mit TRIM28-Komplexen an einem Chromatinlocus.
Literatur:
Informationen zur Thematik KRAB-ZNF/TRIM28
- Czerwińska P, Mazurek S, Wiznerowicz M. The complexity of TRIM28 contribution to cancer. J Biomed Sci. 2017 Aug 29;24(1):63. doi: 10.1186/s12929-017-0374-4. Review. PMID: 28851455
- Ecco G, Imbeault M, Trono D. KRAB zinc finger proteins. Development. 2017 Aug 1;144(15):2719-2729. doi: 10.1242/dev.132605. Review. PMID: 28765213
- Cheng CT, Kuo CY, Ann DK. KAPtain in charge of multiple missions: Emerging roles of KAP1. World J Biol Chem. 2014 Aug 26;5(3):308-20. doi:10.4331/wjbc.v5.i3.308. Review. PMID: 25225599
- Lupo A, Cesaro E, Montano G, Zurlo D, Izzo P, Costanzo P. KRAB-Zinc Finger Proteins: A Repressor Family Displaying Multiple Biological Functions. Curr Genomics. 2013 Jun;14(4):268-78. doi: 10.2174/13892029113149990002. PMID: 24294107
- Iyengar S, Farnham PJ. KAP1 protein: an enigmatic master regulator of the genome. J Biol Chem. 2011 Jul 29;286(30):26267-76. doi: 10.1074/jbc.R111.252569. Review. PMID: 21652716
Ausgewählte eigene Arbeiten
- Al Chiblak M, Steinbeck F, Thiesen HJ, Lorenz P. DUF3669, a "domain of unknown function" within ZNF746 and ZNF777, oligomerizes and contributes to transcriptional repression. BMC Mol Cell Biol. 2019 Dec 19;20(1):60. doi: 10.1186/s12860-019-0243-y. PMID: 31856708
- Born N, Thiesen HJ, Lorenz P. The B-subdomain of the Xenopus laevis XFIN KRAB-AB domain is responsible for its weaker transcriptional repressor activity compared to human ZNF10/Kox1. PLoS One. 2014 Feb 3;9(2):e87609. doi:10.1371/journal.pone.0087609. eCollection 2014. PMID: 24498343
- Lorenz P, Dietmann S, Wilhelm T, Koczan D, Autran S, Gad S, Wen G, Ding G, Li Y, Rousseau-Merck MF, Thiesen HJ. The ancient mammalian KRAB zinc finger gene cluster on human chromosome 8q24.3 illustrates principles of C2H2 zinc finger evolution associated with unique expression profiles in human tissues. BMC Genomics. 2010 Mar 26;11:206. doi: 10.1186/1471-2164-11-206. PMID: 20346131 Free PMC article.
- Ding G, Lorenz P, Kreutzer M, Li Y, Thiesen HJ. SysZNF: the C2H2 zinc finger gene database. Nucleic Acids Res. 2009 Jan;37(Database issue):D267-73. doi: 10.1093/nar/gkn782. PMID: 18974185 Free PMC article.
- Lorenz P, Koczan D, Thiesen HJ. Transcriptional repression mediated by the KRAB domain of the human C2H2 zinc finger protein Kox1/ZNF10 does not require histone deacetylation. Biol Chem. 2001 Apr;382(4):637-44. doi: 10.1515/BC.2001.075. PMID: 11405226
Ansprechpartner
Dr. rer. nat. Peter Lorenz
Institut für Immunologie
Schillingallee 70, 18057 Rostock
peter.lorenz{bei}med.uni-rostock.de
0381 494 - 5879
